一、实验目的
学习DBSCAN算法基本原理,掌握算法针对不同形式数据如何进行模型输入,并结合可视化工具对最终聚类结果开展分析。
二、实验内容
3:使用DBSCAN算法对flower_data数据集进行聚类算法应用。
三、实验结果与分析
【iris数据集的聚类】
在任务1中,需要分别对Sepal和Petal进行聚类。此处使用【sklearn】库中的DBSCAN封装包进行调用,选定初始参数eps = 0.5(领域的半径)、min_samples = 3(领域内最少包括的同类数据个数),采用fit方法进行模型训练,最后得到训练标签为【dbscan_sepal.labels_】和【dbscan_petal.labels_】。整体代码如下图所示。

2:绘出聚类前后的图
在任务2中,定义了图的大小和4个子图,分别用于显示花萼聚类前、花萼聚类后、花瓣聚类前、花瓣聚类后的聚类散点图,以便显示DBSCAN算法聚类前后的效果。整体代码如下图所示。
【子图1:原始的Sepal散点图】

【子图2:聚类后的Sepal散点图】

【子图3:原始的Petal散点图】

【子图4:聚类后的Petal散点图】

程序输出结果如下图所示。与K-means算法相比,数据集采用DBSCAN算法聚类后,Sepal的类别个数为1,Petal的类别个数为2。因为DBSCAN是根据密度进行聚类且无法设置预期的聚类类别个数,同时Sepal中整个数据点的分布是较为均匀的,因此Sepal的最终聚类个数为1;而Petal中整个数据点的分布主要集中在左下角和右上角,因此Petal的最终聚类个数为2。

3:计算并输出轮廓系数和ARI
在任务3中,利用【from sklearn.metrics import silhouette_score】和【from sklearn.metrics import adjusted_rand_score】从评价指标中调用轮廓系数和ARI,输入数据集本身的特征值和DBSCAN算法聚类得到的标签,进行对比后输出轮廓系数和ARI的结果。整体代码如下图所示。

程序输出结果如下图所示。可以看到DBSCAN算法计算出来的花萼聚类的轮廓系数为0.43左右、花瓣聚类的轮廓系数为0.77左右。同时,DBSCAN算法计算出来的花萼聚类的ARI为0.00左右、花瓣聚类的ARI为0.57左右。而在上一次的实验中,K-means算法计算出来的花萼聚类的ARI为0.60左右、花瓣聚类的ARI为0.89左右。

综上所述,针对iris数据集而言,K-means算法的表现比DBSCAN算法的表现更好,因为K-means算法的ARI明显高于DBSCAN算法的ARI。
【blob数据集的聚类】
0:输出数据集的基本信息
在读入数据集的文本文件后,直接打印每一行的特征信息。整体代码如下图所示。

程序的输出结果如下图所示。其中第一列是特征1,第二列是特征2,第三列是类别标签。

在任务1中,需要对blob数据集进行聚类。此处使用【sklearn】库中的DBSCAN封装包进行调用,选定初始参数eps = 0.3(领域的半径)、min_samples = 10(领域内最少包括的同类数据个数),采用fit方法进行模型训练,最后得到训练标签为【dbscan_labels】。整体代码如下图所示。

在任务2中,定义了图的大小和2个子图,分别用于显示blob数据集聚类前、聚类后的散点图,以便显示DBSCAN算法聚类前后的效果。整体代码如下图所示。

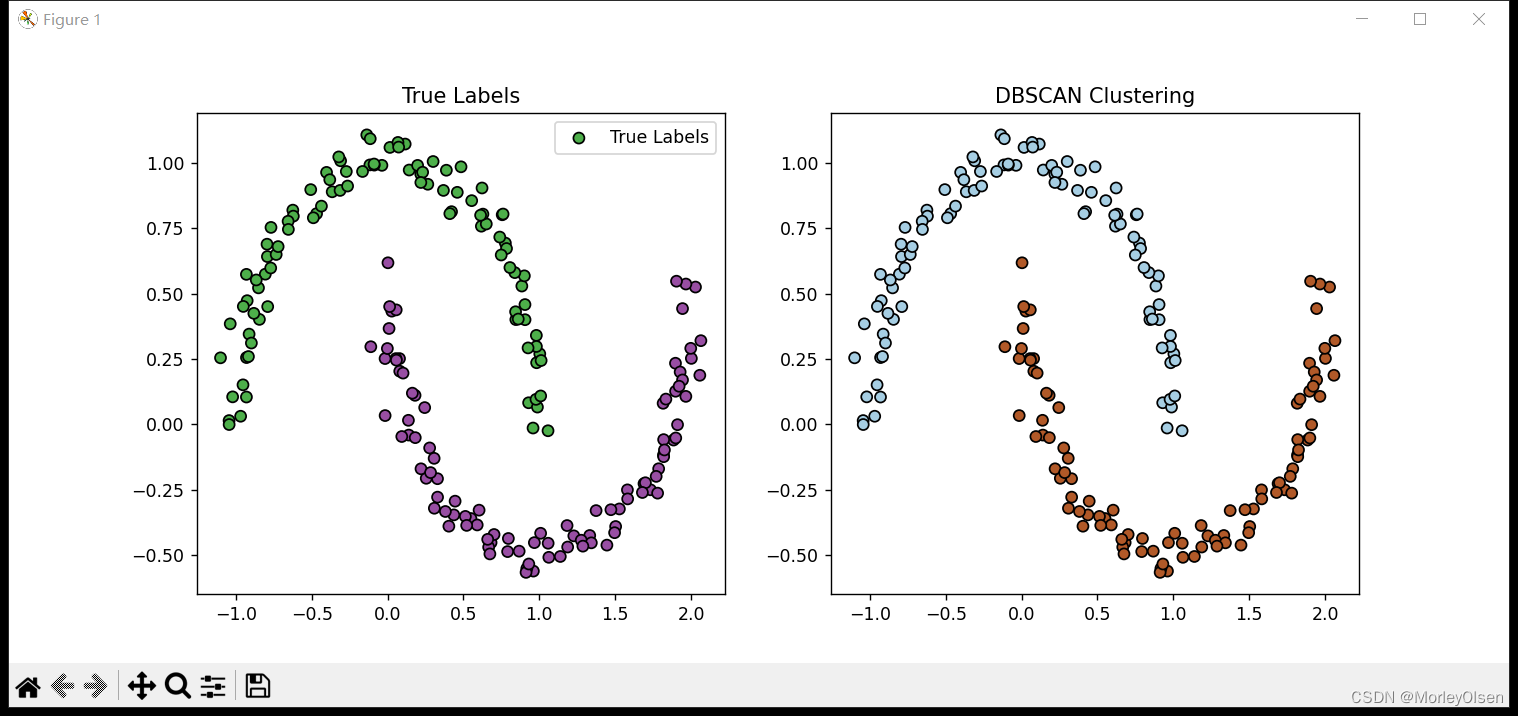
程序输出结果如下图所示。其中,True Labels对应原始blob数据,DBSCAN Clustering对应采用DBSCAN算法聚类后的blob数据。
3:调用K-means进行聚类
在任务3中,需要对blob数据集进行聚类。此处使用【sklearn】库中的KMeans封装包进行调用,选定初始的聚类数目为2,采用fit方法进行模型训练,最后得到训练标签为【kmeans.labels_】。整体代码如下图所示。

4:绘出K-means算法聚类前后的图
在任务4中,定义了图的大小和2个子图,分别用于显示blob数据集聚类前、聚类后的散点图,以便显示DBSCAN算法聚类前后的效果。整体代码如下图所示。

程序输出结果如下图所示。其中,True Labels对应原始blob数据,KMeans Clustering对应采用K-means算法聚类后的blob数据。

5:分别计算并输出K-means算法和DBSCAN算法聚类后的轮廓系数
在任务5中,利用【from sklearn.metrics import silhouette_score】从评价指标中调用轮廓系数,输入数据集本身的标签和聚类算法得到的标签,进行对比后输出轮廓系数的结果。整体代码如下图所示。

程序输出结果如下图所示。可以看到计算出来的K-means算法聚类后的轮廓系数为0.48左右,而DBSCAN算法聚类后的轮廓系数为0.32左右。

6:分别计算并输出K-means算法和DBSCAN算法聚类后的Calinski-Harabasz指数
在任务6中,利用【from sklearn.metrics import calinski_harabasz_score】从评价指标中调用Calinski-Harabasz指数,输入数据集本身的标签和聚类算法得到的标签,进行对比后输出Calinski-Harabasz指数的结果。整体代码如下图所示。

程序输出结果如下图所示。可以看到计算出来的K-means算法聚类后的Calinski-Harabasz指数为291.22左右,而DBSCAN算法聚类后的Calinski-Harabasz指数为127.23左右。

7:分别计算并输出K-means算法和DBSCAN算法聚类后的ARI
在任务7中,利用【from sklearn.metrics import adjusted_rand_score】从评价指标中调用ARI,输入数据集本身的标签和聚类算法得到的标签,进行对比后输出ARI的结果。整体代码如下图所示。

程序输出结果如下图所示。可以看到计算出来的K-means算法聚类后的ARI为0.22左右,而DBSCAN算法聚类后的ARI为1.0。

综上所述,针对blob数据集而言,DBSCAN算法的表现比K-means算法的表现更好,因为K-means算法的ARI明显低于DBSCAN算法的ARI。
【flower_data数据集的聚类】
0:数据集的文件结构
如下图所示,数据集根目录下共分为5类图像,分别是daisy、dandelion、roses、sunfls、tulips。

1:向程序导入数据集图像
在任务1中,首先利用dadaset_path变量存储数据集的路径,然后利用循环遍历每一个根据类别创建的子文件夹,利用cv2.imread读取图像,并将非空的子文件夹的名称添加到label中。整体代码如下图所示。

2:提取图像特征
在任务2中,定义使用色彩直方图作为图像的特征并提取特征的函数extract_color_histogram(),并让存储图像的列表image_list中的每一张图片调用该特征提取函数。整体代码如下图所示。

3:数据标准化
在任务3中,使用numpy中的array,对提取出来的特征向量进行标准化操作。整体代码如下图所示。

4:使用PCA降维
在任务4中,由于图像的特征通道非常多,而要在二维聚类散点图中体现数据的分布,所以需要将原始的图像特征维数降低至2。整体代码如下图所示。

5:采用K-means算法进行聚类
在任务5中,需要对flower数据集进行聚类。此处使用【sklearn】库中的KMeans封装包进行调用,选定初始的聚类数目为5,采用fit方法进行模型训练,最后得到训练标签为【kmeans.labels_】。整体代码如下图所示。

在任务6中,需要对flower数据集进行聚类。此处使用【sklearn】库中的DBSCAN封装包进行调用,选定初始参数eps = 0.1(领域的半径)、min_samples = 50(领域内最少包括的同类数据个数),采用fit方法进行模型训练,最后得到训练标签为【kmeans.labels_】。整体代码如下图所示。

在任务6中,定义了图的大小,并定义了3个子图,分别用于显示聚类前、采用K-means算法聚类后、采用DBSCAN算法聚类后的散点图。整体代码如下图所示。
【子图1:原始数据的散点图】

【子图2:采用K-means算法聚类后散点图】


程序输出结果如下图所示。可以发现K-Means算法将数据点切成了5块(主要依靠降维后的2个特征进行分割),而DBSCAN算法根据密度将数据点分成了2块(高密度为红色,低密度为紫色)。

四、遇到的问题和解决方法
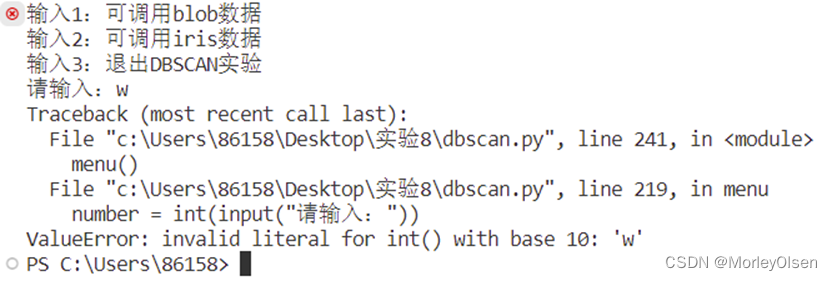
问题1:在一开始对菜单功能采用整型变量接受用户输入的信息的方法进行设定的时候,如果用户输入非数字的信息,则程序会出现报错,即无法将str信息成功转换为int信息。程序部分代码如图1所示,程序报错信息如图2所示。

解决1:将接收信息的number变量直接定义为string类型,然后在if-else判断组语句中,将数字也变为字符格式。修改的程序部分代码如图1所示,程序完整性检测如图2所示。


问题2:在处理flower数据集的时候,画图中无法将标签进行读取。具体报错如下图所示。

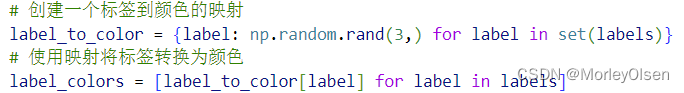
解决2:在画图之前建立标签映射,把每个类别的字符串标签映射到一种颜色中。具体代码如下图所示。
五、实验总结
1:DBSCAN算法的特点主要分为以下几个方面。
- 基于数据点的密度进行聚类:DBSCAN是一种基于数据点密度的聚类算法,不需要事先指定聚类的数量。它通过定义一个密度阈值来确定邻域内的数据点数量,从而将数据点分为核心点、边界点和噪声点。
- 能够发现任意形状的聚类:与K均值等算法不同,DBSCAN能够找到各种形状的聚类,包括非凸形状的聚类。
- 噪声容忍性:DBSCAN能够有效地处理噪声数据点,将它们识别为噪声点而不将其分配给任何聚类。
- 无需事先指定聚类数量:DBSCAN不需要用户事先指定聚类的数量,因为它根据数据的密度自动确定聚类的数量。
- 适用于不均匀分布的数据:DBSCAN适用于不同密度的数据集,能够有效地识别密集区域和稀疏区域中的聚类。
- 簇的连接性:DBSCAN通过将具有足够密度的核心点连接起来来形成聚类,从而保持了簇内数据点的连接性。
2:DBSCAN算法和K-means算法的区别主要分为以下几个方面。
- 聚类方式:K-means算法将数据点分成K个互不重叠的簇,每个簇的中心由簇内数据点的均值计算得出。K均值需要事先指定聚类的数量K。DBSCAN算法不需要事先指定聚类数量,而是通过定义邻域的方式,将数据点划分为核心点、边界点和噪声点。
- 对噪声的处理:K-means算法对噪声数据点敏感,离其他簇很远的噪声点也会分配到某一个簇。DBSCAN算法能够有效处理噪声数据点,不将簇外的噪声点分配给任何聚类。
- 聚类形状:K-means算法假定聚类是球状的,每个簇的中心是数据点的平均值,因此对于非球形簇效果不佳。DBSCAN算法由于自身基于数据点的密度的特点,所以能够发现不同形状的聚类,包括非凸形状的聚类。
六、程序源代码
【1:iris数据集 + blob数据集】
| import matplotlib.pyplot as plt from sklearn import datasets from sklearn.cluster import DBSCAN from sklearn.cluster import KMeans import numpy as np import pandas as pd from sklearn.metrics import silhouette_score from sklearn.metrics import calinski_harabasz_score from sklearn.metrics import adjusted_rand_score def print_data(want_print, print_iris): """ 展示iris的数据 :return: None """ print("iris{0}为:\n{1}".format(want_print, print_iris)) print("=" * 85) def blob(): color_list = ['#4daf4a', '#984ea3', '#dede00'] # blobs = datasets.make_moons(n_samples=200, noise=0.05, random_state=42) blobs = np.loadtxt(r'C:\Users\86158\Desktop\blobs.txt')
# 打印整个blob数据 print("Blob的数据如下所示:") for row in blobs: print(row)
X = blobs[:,:2] y = blobs[:,-1].astype(int) color_y = [color_list[y[i]] for i in range(len(X))] # 调用Scikit-learn函数库中的DBSCAN模块进行聚类 dbscan = DBSCAN(eps=0.3, min_samples=10)
# 使用Blobs.txt数据(也可以使用上次的文本和图片数据) dbscan_labels = dbscan.fit_predict(X) # 绘出聚类前后的图 plt.figure(figsize=(12, 5)) """before clustering""" plt.subplot(1, 2, 1) plt.scatter(X[:, 0], X[:, 1], c=color_y, marker='o', edgecolor='k', s=40, label='True Labels') plt.title('True Labels') plt.legend() """after clustering""" plt.subplot(1, 2, 2) plt.scatter(X[:, 0], X[:, 1], c=dbscan_labels, marker='o', edgecolor='k', s=40, cmap=plt.cm.Paired) plt.title('DBSCAN Clustering') plt.show()
""" 相同的数据集,与K-means进行比较 即比较两个聚类方法在非球形聚集的数据集上的表现 """
# 调用Scikit-learn函数库中的kmeans模块进行聚类 kmeans = KMeans(n_clusters=2)
# 使用Blobs.txt数据(也可以使用上次的文本和图片数据) kmeans.fit(X) kmeans_labels = kmeans.labels_
# 绘出聚类前后的图 plt.figure(figsize=(12, 5)) """before clustering""" plt.subplot(1, 2, 1) plt.scatter(X[:, 0], X[:, 1], c=color_y, marker='o', edgecolor='k', s=40, label='True Labels') plt.title('True Labels') plt.legend() """after clustering""" plt.subplot(1, 2, 2) plt.scatter(X[:, 0], X[:, 1], c=kmeans_labels, marker='o', edgecolor='k', s=40, cmap=plt.cm.Paired) plt.title('KMeans Clustering') plt.show()
""" 对二者的metrics进行对比 1:轮廓系数 度量了样本与其所分配的簇之间的相似性和不相似性。 对于每个样本,它计算了与同一簇中其他样本的相似性与与最接近的其他簇中的样本的不相似性之间的差异。 轮廓系数的取值范围在[-1, 1]之间,值越接近1表示聚类效果越好。 2:Calinski-Harabasz指数(方差比准则) 度量了簇内的差异性与簇间的相似性之间的比率。 较高的指数值表示聚类效果较好。 3:ARI(Adjusted Rand Index) 考虑了聚类结果与真实标签之间的一致性。 """ print()
silhouette_kmeans = silhouette_score(X, kmeans_labels) silhouette_dbscan = silhouette_score(X, dbscan_labels) print("轮廓系数比较(Silhouette Score)") print("KMeans:", silhouette_kmeans) print("DBSCAN:", silhouette_dbscan) print() # 对KMeans和DBSCAN的聚类结果计算Calinski-Harabasz指数 calinski_kmeans = calinski_harabasz_score(X, kmeans_labels) calinski_dbscan = calinski_harabasz_score(X, dbscan_labels) print("Calinski-Harabasz指数比较(Calinski-Harabasz Index)") print("KMeans:", calinski_kmeans) print("DBSCAN:", calinski_dbscan) print() # 计算KMeans和DBSCAN的ARI ari_kmeans = adjusted_rand_score(y, kmeans_labels) ari_dbscan = adjusted_rand_score(y, dbscan_labels) print("ARI指数比较(Adjusted Rand Index)") print("KMeans:", ari_kmeans) print("DBSCAN:", ari_dbscan) print() def iris(): # 导入iris数据 iris = datasets.load_iris() # 展示iris真实数据 print_data(want_print="数据", print_iris=iris.data) # 展示iris特征名字 print_data(want_print="特征名字", print_iris=iris.feature_names) # 展示目标值 print_data(want_print="目标值", print_iris=iris.target) # 展示目标值的名字 print_data(want_print="目标值的名字", print_iris=iris.target_names) # 为了便于使用,将iris数据转换为pandas库数据结构,并设立列的名字 # 将iris数据转为pandas数据结构 x = pd.DataFrame(iris.data) # 将iris数据的名字设为‘Sepal_Length’,‘Sepal_Width’,‘Sepal_Width’,‘Petal_Width’ x.columns = ['Sepal_Length', 'Sepal_Width', 'Petal_Length', 'Petal_Width'] # 将iris目标值也转为pandas数据结构 y = pd.DataFrame(iris.target) # 将iris目标值得名字设为‘Targets’ y.columns = ['Targets']
# dbscan for iris
dbscan_sepal = DBSCAN(eps=0.5, min_samples=3) dbscan_sepal.fit(x[['Sepal_Length', 'Sepal_Width']]) x['Sepal_DBSCAN_Cluster'] = dbscan_sepal.labels_ dbscan_petal = DBSCAN(eps=0.5, min_samples=3) dbscan_petal.fit(x[['Petal_Length', 'Petal_Width']]) x['Petal_DBSCAN_Cluster'] = dbscan_petal.labels_ plt.figure(figsize=(16, 7)) plt.subplot(2, 2, 1) plt.scatter(x['Sepal_Length'], x['Sepal_Width'], c=colormap[y['Targets']], s=40, label='Original Data') plt.title('Original Sepal Data') plt.subplot(2, 2, 2) for cluster in np.unique(x['Sepal_DBSCAN_Cluster']): if cluster == -1: # -1 表示噪声点 cluster_data = x[x['Sepal_DBSCAN_Cluster'] == cluster] plt.scatter(cluster_data['Sepal_Length'], cluster_data['Sepal_Width'], c='gray', s=40, label='Noise') else: cluster_data = x[x['Sepal_DBSCAN_Cluster'] == cluster] plt.scatter(cluster_data['Sepal_Length'], cluster_data['Sepal_Width'], c=colormap[cluster], s=40, label=f'Cluster {cluster}') plt.title('Sepal DBSCAN Clustering Overlay') plt.legend() plt.subplot(2, 2, 3) plt.scatter(x['Petal_Length'], x['Petal_Width'], c=colormap[y['Targets']], s=40, label='Original Data') plt.title('Original Petal Data') plt.subplot(2, 2, 4) for cluster in np.unique(x['Petal_DBSCAN_Cluster']): if cluster == -1: # -1 表示噪声点 cluster_data = x[x['Petal_DBSCAN_Cluster'] == cluster] plt.scatter(cluster_data['Petal_Length'], cluster_data['Petal_Width'], c='gray', s=40, label='Noise') else: cluster_data = x[x['Petal_DBSCAN_Cluster'] == cluster] plt.scatter(cluster_data['Petal_Length'], cluster_data['Petal_Width'], c=colormap[cluster], s=40, label=f'Cluster {cluster}') plt.title('Petal DBSCAN Clustering Overlay') plt.legend() plt.tight_layout() plt.show() # 计算轮廓系数 print() print("Iris数据集采用DBSCAN算法聚类的轮廓系数:") # 计算花萼聚类的轮廓系数 silhouette_sepal_dbscan = silhouette_score(x[['Sepal_Length', 'Sepal_Width']], x['Sepal_DBSCAN_Cluster']) print("Sepal:", silhouette_sepal_dbscan) # 计算花瓣聚类的轮廓系数 silhouette_petal_dbscan = silhouette_score(x[['Petal_Length', 'Petal_Width']], x['Petal_DBSCAN_Cluster']) print("Petal:", silhouette_petal_dbscan)
# 计算调整兰德系数 print() print("Iris数据集采用DBSCAN算法聚类的ARI:") # 计算花萼聚类的ARI ari_sepal_dbscan = adjusted_rand_score(iris.target, x['Sepal_DBSCAN_Cluster']) print("Sepal:", ari_sepal_dbscan) # 计算花瓣聚类的ARI ari_petal_dbscan = adjusted_rand_score(iris.target, x['Petal_DBSCAN_Cluster']) print("Petal:", ari_petal_dbscan) print()
def menu(): while 1: print("请根据提示输入这次实验时所调入的数据") print("输入1:可调用blob数据") print("输入2:可调用iris数据") print("输入3:退出DBSCAN实验")
number = input("请输入:") if number == '1': # blob数据使用dbscan blob() elif number == '2': # iris数据使用dbscan iris() elif number == '3': # 退出系统 break else: print("输入数字错误,请重新输入") print()
print("谢谢使用") print() if __name__ == '__main__': # 创建色板图 colormap = np.array(['red', 'lime', 'black']) menu() |
【2:flower_data数据集】
| import numpy as np import cv2 from sklearn.cluster import DBSCAN, KMeans from sklearn.decomposition import PCA import matplotlib.pyplot as plt import os # 1. 准备数据和图像 dataset_path = r'C:\Users\86158\Desktop\flower_photos' # 数据集目录路径 image_list = [] # 用于存储图像的列表 labels = [] # 用于存储标签的列表 for root, dirs, files in os.walk(dataset_path): for filename in files: if filename.endswith('.jpg'): # 仅处理.jpg文件 image_path = os.path.join(root, filename) label = os.path.basename(root) # 获取类别标签 image = cv2.imread(image_path) # 读取图像 if image is not None: image_list.append(image) labels.append(label) # 2. 提取图像特征 # 使用色彩直方图作为特征 def extract_color_histogram(image, bins=(8, 8, 8)): hsv_image = cv2.cvtColor(image, cv2.COLOR_BGR2HSV) hist = cv2.calcHist([hsv_image], [0, 1, 2], None, bins, [0, 180, 0, 256, 0, 256]) hist = cv2.normalize(hist, hist).flatten() return hist # 提取特征向量 features = [] for img in image_list: hist = extract_color_histogram(img) features.append(hist) # 3. 数据标准化 X = np.array(features) # 4. 使用PCA降维 n_components = 2 # 设置PCA降维后的维度 pca = PCA(n_components=n_components) X_pca = pca.fit_transform(X) # 5. 聚类前的散点图按照标签画出 # 创建一个标签到颜色的映射 label_to_color = {label: np.random.rand(3,) for label in set(labels)} # 使用映射将标签转换为颜色 label_colors = [label_to_color[label] for label in labels] plt.figure(figsize=(12, 5)) plt.subplot(1, 3, 1) plt.scatter(X_pca[:, 0], X_pca[:, 1], c=label_colors, cmap='rainbow', marker='o', s=25) plt.title('Scatter Plot Before Clustering (PCA)') kmeans = KMeans(n_clusters=5) kmeans.fit(X_pca) cluster_labels_kmeans = kmeans.labels_ # 7. 聚类后的散点图按照标签画出 plt.subplot(1, 3, 2) plt.scatter(X_pca[:, 0], X_pca[:, 1], c=cluster_labels_kmeans, cmap='rainbow', marker='o', s=25) plt.title('K-means Clustering (PCA)') dbscan = DBSCAN(eps=0.1, min_samples=50) cluster_labels_dbscan = dbscan.fit_predict(X_pca) # 9. 聚类后的散点图按照标签画出 plt.subplot(1, 3, 3) plt.scatter(X_pca[:, 0], X_pca[:, 1], c=cluster_labels_dbscan, cmap='rainbow', marker='o', s=25) plt.title('DBSCAN Clustering (PCA)') plt.tight_layout() plt.show() |