1.相关概念
DBSCAN是基于密度的聚类算法,该类算法假设聚类结构能够通过样本分布的紧密程度确定(样本密度均匀分布),它通常考虑的是样本之间的可连接性,并以最大连接性确定聚类簇。要搞懂该算法,首先要理清楚几个概念:
- 邻域:对于样本 x i ∈ D x_i \in D xi∈D,其邻域包含样本集D中距离 x i x_i xi不超过 ϵ \epsilon ϵ的样本,即 N ϵ ( x i ) = { x j ∈ D ∣ d i s t ( x i , x j ) ≤ ϵ } N_\epsilon(x_i)=\{x_j \in D | dist(x_i,x_j) \leq \epsilon \} Nϵ(xi)={xj∈D∣dist(xi,xj)≤ϵ}。若采用欧式距离,那么 N ϵ ( x i ) N_\epsilon(x_i) Nϵ(xi)就是以 x i x_i xi为圆心,以 ϵ \epsilon ϵ为半径的圆域。
- 核心对象: x i x_i xi的邻域中至少包含 m i n P t s minPts minPts个样本,即 ∣ N ϵ ( x i ) ∣ ≥ m i n P t s |N_\epsilon(x_i)| \geq minPts ∣Nϵ(xi)∣≥minPts,则 x i x_i xi是一个核心对象。这说明核心对象紧邻着多个样本,所以核心对象是算法关注的对象。
- 密度直达: x j x_j xj在 x i x_i xi的邻域中且 x i x_i xi是核心对象,则称 x j x_j xj由 x i x_i xi密度直达,记作 x i → x j x_i \rightarrow x_j xi→xj。不难理解,因为核心对象 x i x_i xi与邻域中的样本紧密的挨着,我们可以认为 x i x_i xi和 x j x_j xj是直接可达的。
- 密度可达:对于样本 x i x_i xi和 x j x_j xj,若其间存在密度直达序列 p 1 , p 2 , . . . , p n p_1,p_2,...,p_n p1,p2,...,pn,其中 p 1 = x i , p n = x j p_1 = x_i,p_n=x_j p1=xi,pn=xj,则称 x j x_j xj由 x i x_i xi 密度可达。例如, x 1 → x 2 → x 3 → x 4 x_1 \rightarrow x_2 \rightarrow x_3 \rightarrow x_4 x1→x2→x3→x4,则称 x 4 由 x 1 x_4由x_1 x4由x1密度可达。而且 x 1 , x 2 , x 3 x_1,x_2, x_3 x1,x2,x3一定是核心对象, x 4 x_4 x4不一定。这说明,我们访问核心对象领域中的样本,就能访问到下一个核心对象,直至邻域中不再含有核心对象,这样我们能访问到所有可达的核心对象及其邻域中的样本。
- 密度相连:若
x
i
和
x
j
x_i和x_j
xi和xj间存在样本
x
k
x_k
xk,使得
x
i
和
x
j
x_i和x_j
xi和xj均由
x
k
x_k
xk密度可达,称
x
i
和
x
j
x_i和x_j
xi和xj密度相连。注意,密度可达一定密度相连。
上图给出了这些概念的示意图。其中, x 1 x_1 x1和 x 2 x_2 x2是核心对象, x 2 x_2 x2由 x 1 x_1 x1密度直达, x 3 x_3 x3由 x 1 x_1 x1密度可达, x 3 x_3 x3和 x 4 x_4 x4密度相连。基于这些概念,DBSCAN将簇定义为:由密度可达推导出最大密度相连样本的集合。 翻译成数学语言:
- 连接性: 若 x i , x j ∈ C 若x_i,x_j \in C 若xi,xj∈C,则 x i x_i xi与 x j x_j xj密度相连。
- 最大性: x i ∈ C x_i \in C xi∈C且 x j x_j xj由 x i x_i xi密度可达,则 x j ∈ C x_j \in C xj∈C。
即同一簇内任意两点密度相连,分属不同簇的任意两点不可能密度可达。
2.算法流程
DBSCAN的思想:先找到样本中所有核心对象,再从任意核心对象出发,逐个访问其邻域的样本找到下一个核心对象及其邻域,如此迭代下去能访问到所有密度可达的样本,这样构成的集合满足连接性和最大性是一个簇,所有核心对象都访问过之后能划分出所有簇。具体流程如下:
- 输入:样本集D和邻域参数( ϵ , M i n P t s \epsilon,MinPts ϵ,MinPts)
- 初始化核心对象集合 Ω = ∅ \Omega = \varnothing Ω=∅
- for i =1,2,3,…,m do
- \enspace\enspace 找到 x i x_i xi的邻域 N ϵ ( x i ) N_\epsilon(x_i) Nϵ(xi)
- \enspace\enspace if ∣ N ϵ ( x i ) ∣ ≥ m i n P t s |N_\epsilon(x_i)| \geq minPts ∣Nϵ(xi)∣≥minPts then
- \enspace\enspace\enspace 将核心对象 x i x_i xi添加到 Ω \Omega Ω
- \enspace\enspace end if
- end for
- 初始化聚类数:k=0
- 初始化为本访问的样本集合: T = D T = D T=D
- while Ω ≠ ∅ \Omega \neq \varnothing Ω=∅ do
- \enspace\enspace T o l d = T T_{old} = T Told=T
- \enspace\enspace 随机选择一个核心对象 O ∈ Ω O \in \Omega O∈Ω
- \enspace\enspace 初始化一个队列Q = < O >
- \enspace\enspace T = T − O T = T - O T=T−O
- \enspace\enspace while Q ≠ ∅ Q \neq \varnothing Q=∅ do
- \enspace\enspace\enspace 取出对首元素q = Q.get()
- \enspace\enspace\enspace if ∣ N ϵ ( q ) ∣ ≥ m i n P t s |N_\epsilon(q)| \geq minPts ∣Nϵ(q)∣≥minPts then
- \enspace\enspace\enspace\enspace Δ = N ϵ ( q ) ∩ T \Delta = N_\epsilon(q)\cap T Δ=Nϵ(q)∩T
- \enspace\enspace\enspace\enspace 将 Δ \Delta Δ的样本加入队列Q
- \enspace\enspace\enspace\enspace T = T − Δ T=T-\Delta T=T−Δ
- \enspace\enspace\enspace end if
- \enspace\enspace end while
- \enspace\enspace k=k+1
- \enspace\enspace 生成簇为访问过的样本 C k = T o l d − T C_k =T_{old}-T Ck=Told−T
- \enspace\enspace 删除访问过的核心对象 Ω = Ω − C k \Omega =\Omega -C_k Ω=Ω−Ck
- end while
- 输出:所有簇
算法瓶颈:DBSCAN的瓶颈在于求邻域 N ϵ ( x i ) N_\epsilon (x_i) Nϵ(xi),因为每次都要计算所有样本与 x i x_i xi的距离,当样本规模很大时求邻域相当费时。可以采用网格的方法,大量减少访问的样本数。
python_52">3.python实现
python">#确定x的领域
def neiborgh(data,x,epsilon):
indexs = []
for i in range(0,data.shape[0]):
dist = math.sqrt((data[i]-x)@(data[i]-x).T)
if dist <= epsilon:
indexs.append(i)
return data[indexs]
#求二维数组的交集
def intersect(A,B):
lows=[]
for i in range(0,A.shape[0]):
if (B == A[i]).all(1).any():
lows.append(i)
return A[lows]
#求二维数组的差集
def difference(A,B):
inter = intersect(A,B)
lows = []
for i in range(0,A.shape[0]):
if (inter == A[i]).all(1).any():
lows.append(i)
return np.delete(A,lows,axis = 0)
def dbscan(data,epsilon,minPts):
m,n = data.shape
#初始化核心对象集合
centers = np.zeros((1,n))
#寻找所有核心对象
for i in range(0,m):
N = neiborgh(data,data[i],epsilon)
if N.shape[0] >= minPts:
centers = np.concatenate((centers,[data[i]]),axis=0)#行拼接
#删除第一行
centers = np.delete(centers,0,0)
#簇的个数
k = 0
#未被访问的集合
I = data
#初始化簇的集合
cls =[]
#外层循环随机选择一个核心对象为出发点
while centers.shape[0]!=0:
Iold = I.copy()
#随机选择一个核心对象,加入队列
r = np.random.randint(0,centers.shape[0])
Q = Queue(maxsize=0)
Q.put(centers[r])
I = difference(I,[centers[r]])
#内层循环找到所有相连的核心对象,及其领域
while Q.qsize() > 0:
q = Q.get()
N =neiborgh(data,q,epsilon)
#如果是核心对象的话
if N.shape[0] >= minPts:
ins = intersect(I,N)
#交集中所有样本加入队列
for d in ins:
Q.put(d)
I = difference(I,ins)
k=k+1
#访问过的核心对象及其领域,形成一簇
ck = difference(Iold,I)
cls.append(ck)
#删去访问过的核心对象
centers = difference(centers,ck)
return cls
随机生成三簇数据检验DBSCAN聚类效果:
python">def showClusters(c1,c2,c3):
plt.scatter(c1[:,0],c1[:,1],c='r',s=10)
plt.scatter(c2[:,0],c2[:,1],c='b',s=10)
plt.scatter(c3[:,0],c3[:,1],c='g',s=10)
plt.show()
moids = [[2,2],[8,2],[0,8]]
X = datasets.make_blobs(n_samples=200, n_features=2, centers=moids,cluster_std=1)[0]
#X=datasets.make_classification(n_samples=200,n_features=2,n_informative=2,n_redundant=0,n_repeated=0,n_clusters_per_class=1)[0]
cls = dbscan(X,0.88,4)
plt.scatter(X[:,0],X[:,1],s=10)
plt.show()
showClusters(cls[0],cls[1],cls[2])
原始数据分布和聚类后的效果图如下:
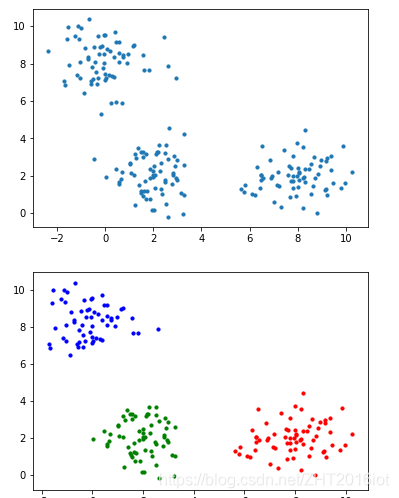
可以看出DBSCAN聚类后簇的分布与原始数据分布基本一致,更重要地是,DBSCAN聚类过后一些离群点被抛弃了,因为离群点与任意核心对象都密度不可达,所以是访问不到的,这是DBSCAN的一个优点。